Con l’aumento della richiesta di sequenziamento del genoma, i ricercatori in questi anni stanno generando una quantità inimmaginabile di informazioni che raddoppia di dimensioni ogni anno.
Questi dati, generati dal sequenziamento dell’intero genoma, richiedono grandi risorse in termini di elaborazione, archiviazione e gestione che possono portare a una significativa riduzione delle prestazioni.
Per stare al passo con l’aumento della domanda di sequenziamento, le organizzazioni devono quindi essere in grado di completare più processi di sequenziamento in meno tempo senza sacrificare precisione o sicurezza.
Alcuni numeri. Risale oramai a 15 anni fa il sequenziamento del primo genoma umano. Ci si lavorava da quasi 20 anni ed è costato circa 3 miliardi di dollari. Oggi è possibile sequenziare 3 miliardi di paia di basi in meno di un giorno e un costo inferiore ai 1.000 euro, rendendo possibile l’utilizzo del sequenziamento dell’intero genoma per ricerche ed applicazioni cliniche.
Tra le aziende che dispongono delle tecnologie software necessarie per questo compito, Parabricks sfrutta un software di analisi genomica con accelerazione in hardware, a livello di Gpu, ad alte prestazioni. Così riesce ad accelerare i tempi di elaborazione delle pipeline genomiche.
Con gli approcci basati sulle Cpu standard, l’elaborazione potrebbe richiedere giorni, ma con le soluzioni di Parabricks bastano pochi minuti. Il software Parabricks lavora in combinazione con l’architettura NetApp Ontap AI, supportata dai supercomputer Nvidia Dgx e dallo storage NetApp connesso al cloud. Nel complesso parliamo quindi di una soluzione AI end-to-end integrata e ottimizzata per l’elaborazione di dati genomici.
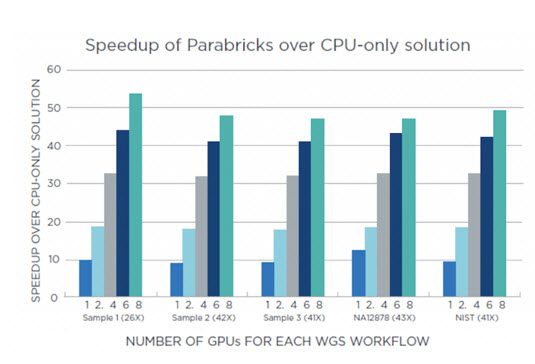
Con la soluzione congiunta, a partire dalle tecnologie di NetApp e Parabricks è possibile accelerare il sequenziamento del genoma in media di 50 volte, rispetto alle soluzioni che sfruttano solo la Cpu, ma anche eseguire più operazioni di analisi in meno tempo grazie allo storage all-flash connesso al cloud da NetApp e Nvidia.
La soluzione permette di ridurre ridurre il costo di proprietà con le tecnologie di efficienza dei dati e ottenere una capacità 25 volte maggiore rispetto a sistemi concorrenti. Significa anche un ritorno degli investimenti sicuro reso possibile dall’integrazione e dall’automazione dell’orchestrazione dei dati in cloud con quelli on-premise.
Nel test del grafico appena sopra sono stati utilizzati Ontap AI con un controller di archiviazione Aff A800 NetApp, otto Gpu Nvidia Tesla V100 Sxm2 da 32 Gbyte, Ubuntu 16.04 e 526 Gbyte di memoria.
In sintesi, Parabricks è riuscita ad ottimizzare il throughput per completare più processi di elaborazione genomica in meno tempo.
Questa soluzione integrata offre la flessibilità necessaria per adattarsi a più attività genomiche e soddisfare la domanda consentendo di eliminare le congetture e accelerare il sequenziamento grazie a un’architettura di riferimento convalidata che riduca la complessità del design e faciliti il lavoro.
© RIPRODUZIONE RISERVATA











































